致病菌耐药已成为当前传染病防控的重要问题。世界范围内霍乱弧菌耐药率的不断上升,不同地区报告耐药情况亦较复杂。腹泻病室对我国O1和O139两个主要致病血清群霍乱弧菌菌株的耐药谱进行了研究。其中O1群El Tor型包括了第七次霍乱大流行开始至今(1961年-2010年)的菌株,O139群包括了1993年5月首次传入我国后至今的各年代菌株。结果显示两个血清型流行菌株的耐药谱有很大的不同:(1)O1群El Tor型菌株耐药尚不严重,除奈啶酸、四环素和复方新诺明的耐药率在第三个流行期之后上升到近30%外,其余检测的9种抗生素的敏感率都在95%以上,与其他国家一些监测报道有不同;而O139群菌株基本和国外菌株的报道相似,耐药严重,超过半数的菌株对氯霉素、卡那霉素、萘啶酸、四环素、氨苄西林和复方新诺明耐药。(2)O1群El Tor型菌株中耐药菌株出现于八十年代之后,且多耐药菌株比例很低。而O139群菌株出现流行时即出现多耐药菌株,已逐渐形成一个优势的八重多耐药谱,并在1998-2006年间分离菌株中占较大比例。另外,来自环境水体的非产毒O1群菌株应代表了环境自然菌群,但一些菌株仍具有多耐药特征,这可能是自发突变的积累,但其潜在的威胁是,这些菌株可能通过基因水平转移的方式将耐药性传递给环境中的产毒株。O139群菌株中还显示SXT因子与一些特征性药物抗性有关联。这两项研究结果已分别发表在《International Journal of Antimicrobial Agents》和《PLoS ONE》杂志上。
针对O139群菌株的喹诺酮类耐药,还对O139群霍乱弧菌中拓扑异构酶基因突变诱发细菌对喹诺酮类抗生素耐药进行了研究。作为喹诺酮类药物作用靶位,DNA拓扑异构酶II(编码基因gyrA、gyrB)和IV(编码基因parC、parE)可通过基因突变减弱抗生素的抗菌活性。研究表明87.8%的O139群萘啶酸耐药菌株在GyrA上83位丝氨酸突变为异亮氨酸、ParC上85位丝氨酸突变为亮氨酸,但这些菌株对环丙沙星仍较为敏感。再深入对具有这两个突变的8株环丙沙星耐药菌株进行序列分析,发现均在GyrA第87位天冬氨酸突变为天冬酰胺、ParE第420位天冬氨酸突变为天冬酰胺或者第439位脯氨酸突变为丝氨酸,其中ParE中氨基酸突变在霍乱弧菌中为首次报道。我们通过在萘啶酸耐药菌株中进行定点突变,证明GyrA第87位天冬氨酸突变为天冬酰胺使菌株对环丙沙星耐药性提升4倍,进一步结合ParE上的氨基酸突变可使环丙沙星耐药性提升17倍以上,即两种多位点的突变组合可以使细菌对环丙沙星产生耐药。这些结果发表在《International Journal of Antimicrobial Agents》杂志上,为细菌喹诺酮耐药提供了新的拓扑异构酶基因突变位点和组合突变信息,而且从实验上证明了这些突变的意义。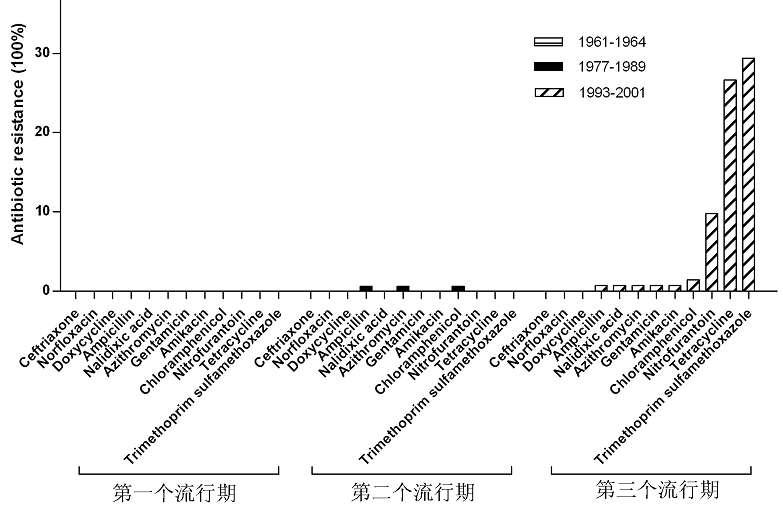
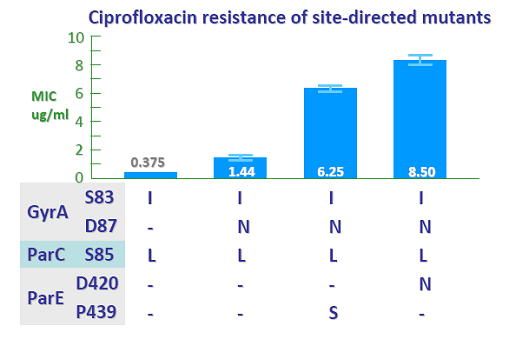
针对O139群菌株的喹诺酮类耐药,还对O139群霍乱弧菌中拓扑异构酶基因突变诱发细菌对喹诺酮类抗生素耐药进行了研究。作为喹诺酮类药物作用靶位,DNA拓扑异构酶II(编码基因gyrA、gyrB)和IV(编码基因parC、parE)可通过基因突变减弱抗生素的抗菌活性。研究表明87.8%的O139群萘啶酸耐药菌株在GyrA上83位丝氨酸突变为异亮氨酸、ParC上85位丝氨酸突变为亮氨酸,但这些菌株对环丙沙星仍较为敏感。再深入对具有这两个突变的8株环丙沙星耐药菌株进行序列分析,发现均在GyrA第87位天冬氨酸突变为天冬酰胺、ParE第420位天冬氨酸突变为天冬酰胺或者第439位脯氨酸突变为丝氨酸,其中ParE中氨基酸突变在霍乱弧菌中为首次报道。我们通过在萘啶酸耐药菌株中进行定点突变,证明GyrA第87位天冬氨酸突变为天冬酰胺使菌株对环丙沙星耐药性提升4倍,进一步结合ParE上的氨基酸突变可使环丙沙星耐药性提升17倍以上,即两种多位点的突变组合可以使细菌对环丙沙星产生耐药。这些结果发表在《International Journal of Antimicrobial Agents》杂志上,为细菌喹诺酮耐药提供了新的拓扑异构酶基因突变位点和组合突变信息,而且从实验上证明了这些突变的意义。












 2013-05-06
2013-05-06
 【打印】
【打印】

