
中国疾病预防控制中心传染病预防控制所、传染病溯源预警与智能决策全国重点实验室和中国科学院微生物研究所共同研究和建立了一个综合性的病原菌质粒基因组数据库(PIPdb,https://nmdc.cn/pipdb/),包含来自 110 个病原菌属的 450 种致病物种的 1,009,571 个基因组的 792,964 个质粒序列片段簇(Plasmid Segment Clusters,PSCs)。目前,PIPdb 是第一个专门针对病原菌的质粒基因组数据库,且提供详细的多维元数据,如质粒的宿主细菌种属、采集时间、地点和生境信息。PIPdb 还提供全面的注释功能,包括质粒MLST、插入序列(IS)、整合子、复制起始位点(oriT)、松弛酶、T4CP、毒力因子、重金属抗性基因和抗生素抗性基因等。该数据库具有友好的用户界面,方便用户对来自不同宿主细菌、分离生境的质粒进行查询。
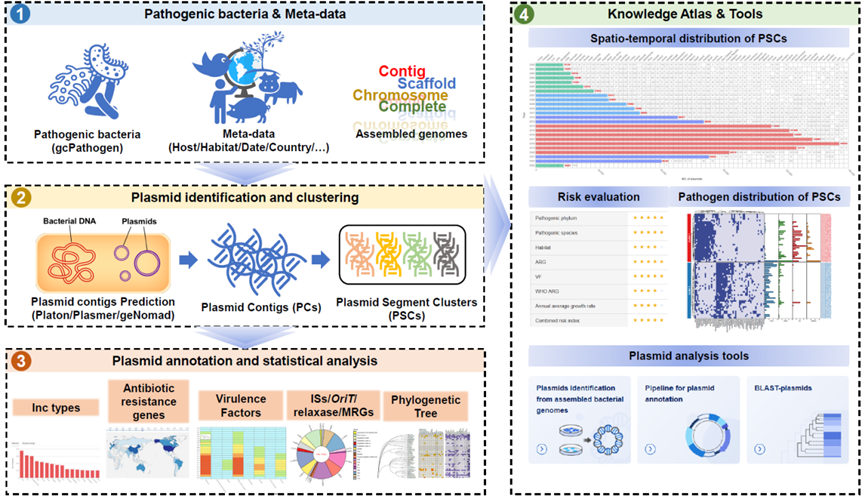
图1 病原菌质粒数据库PIPdb的图片摘要
在PIPdb中,以四种主要类别(宿主细菌种类、耐抗生素类型、毒力因子类型、分离生境信息)对PSCs数据进行了系统性整理(图 2)。用户可根据多维元数据,自主对质粒进行高级查询。基于传染病所微生物组学研究室之前研发的PlasRing在线分析流程二次开发,在PIPdb中还提供了三个易于访问的分析工具接口,用于快速分析质粒。用户可以通过上传 FASTA 格式文件轻松进行质粒序列识别和分析。工具“Plasmid identification from assembled bacterial genomes”能够准确识别组装细菌基因组中的质粒序列片段(contigs)。工具“Pipeline for Plasmid annotation”侧重于质粒序列的详细功能基因注释,能够精确快速地识别质粒特征。工具“BLAST-plasmids”允许用户将其序列与 PIPdb 数据库中的质粒序列进行比对。
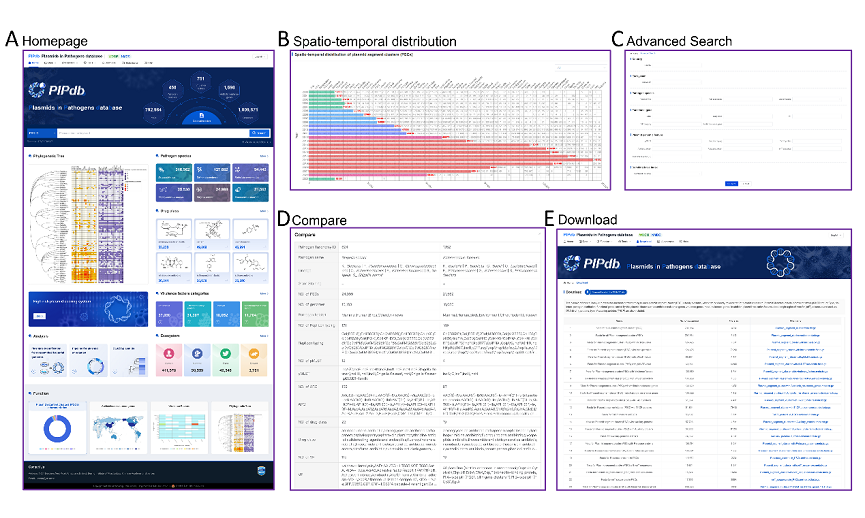
图2 PIPdb的页面
基于PIPdb,研究组进行了一系列病原菌所携带的质粒的汇总分析。与其他生境相比,与“人类”相关的PSCs在病原菌属中分布更为广泛,且每个基因组携带的PSCs数量更为稳定。随着时间的推移,大多数病原菌属的每个基因组平均 PSCs 数量呈现最初增加,近年来有所下降的趋势,特别是在大肠埃希菌、肺炎克雷伯菌、志贺菌和肠球菌等中趋势明显(图 3)。但在梭菌属、拟杆菌属和嗜血杆菌属中则变化较小。
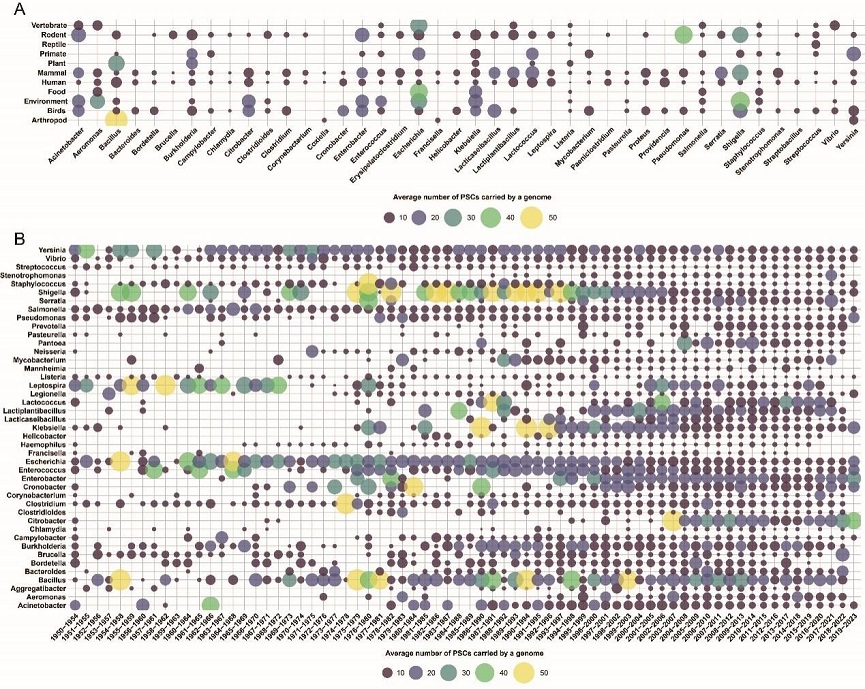
图3 不同生境和不同采样时间的病原菌属中平均每个基因组携带的PSCs数目
对 PSCs 的分析显示,绝大多数(92.96%)局限于单个病原菌属中,但也有小部分(0.01%)的PSCs可在超过15个属中分布。根据这些广泛分布的 PSCs 在不同属中的存在情况,进一步分为两个不同的簇(图 4),显示了质粒分布的宿主种属特征,提示了不同的宿主适应机制。

图4 PSCs分布及宽宿主质粒的基因组特征
通过质粒水平基因转移传播VFGs是另一个需要关注的方面。通过对PIPdb数据归纳分析发现志贺菌属、巴尔通体属、军团菌属和弧菌属中的PSCs主要携带与效应物传递系统相关的VFGs。而肠球菌属、嗜血杆菌属和链球菌属中的大多数PSCs主要携带与黏附相关的VFGs(图 5)。
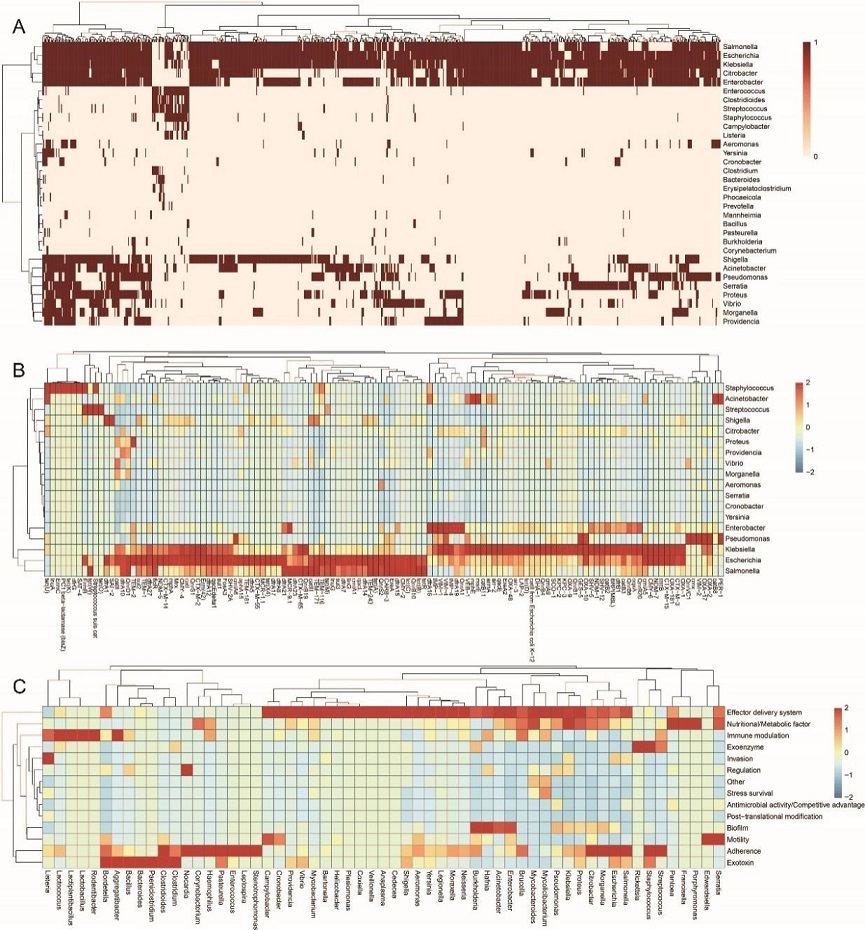
图5 PIPdb中PSCs及其携带的耐药基因和毒力基因类别的分布
PIPdb是专门收集与病原菌相关质粒基因组的资源数据库,是传染病防控One Health框架下病原菌质粒研究的有力的工具,对传染病防控、遏制耐药等公共卫生应用均具有重要价值。数据库将定期持续更新,后期也会接收用户提交的质粒完成图序列,并反馈质粒序列号。
PIPdb为中国疾病预防控制中心传染病所、传染病溯源预警与智能决策全国重点实验室和中国科学院微生物研究所共同构建,以上研究以“PIPdb: a comprehensive plasmid sequence resource for tracking the horizontal transfer of pathogenic factors and antimicrobial resistance genes”为题发表于2024年的Nucleic Acids Research(为JCR分区生化和分子生物学Q1区,影响因子16.6)杂志上。朱倩慧和陈奇并列为本文第一作者,卢昕研究员、吴林寰研究员、胡松年研究员和阚飙研究员同为本文通讯作者。










 2024-12-26
2024-12-26
 【打印】
【打印】

